A phytools user asks:
"I have a list of names that correspond exactly to the tips of a phylogenetic tree. What i'd like to do is obtain the branch lengths that correspond to these tip edges."
This can be done with one line:
n<-length(tree$tip.label)
ee<-setNames(tree$edge.length[sapply(1:n,function(x,y) which(y==x),y=tree$edge[,2])],tree$tip.label)
(Or at least this would be one line if the width of this entry window permitted me to put length(tree$tip.label) into my sapply block instead of first calculating n.)
ee<-setNames(tree$edge.length[sapply(1:n,function(x,y) which(y==x),y=tree$edge[,2])],tree$tip.label)
We get a vector with all the terminal edge lengths with names set to all the tip names in the tree, so we can rearrange or select a subset of these tip lengths on that basis.
Let's check it:
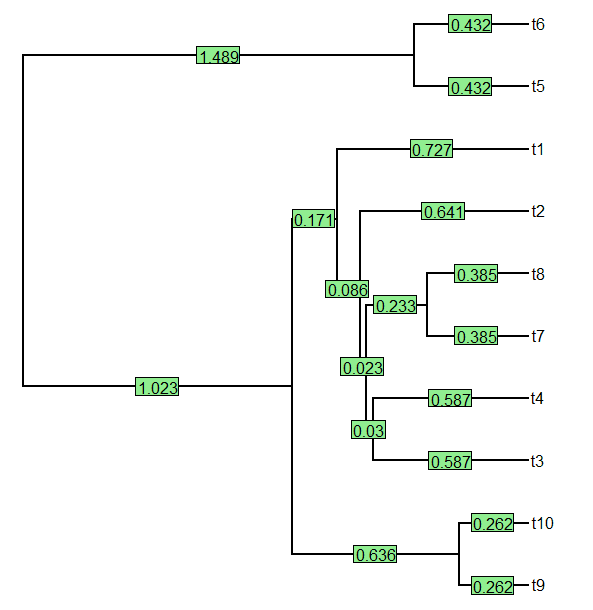
> tree<-pbtree(n=10)
> tree$edge.length<-round(tree$edge.length,3)
> n<-length(tree$tip.label)
> ee<-setNames(tree$edge.length[sapply(1:n,function(x,y) which(y==x),y=tree$edge[,2])],tree$tip.label)
> ee
t9 t10 t3 t4 t7 t8 t2 t1 t5 t6
0.262 0.262 0.587 0.587 0.385 0.385 0.641 0.727 0.432 0.432
> plotTree(tree)
> edgelabels(tree$edge.length)
> tree$edge.length<-round(tree$edge.length,3)
> n<-length(tree$tip.label)
> ee<-setNames(tree$edge.length[sapply(1:n,function(x,y) which(y==x),y=tree$edge[,2])],tree$tip.label)
> ee
t9 t10 t3 t4 t7 t8 t2 t1 t5 t6
0.262 0.262 0.587 0.587 0.385 0.385 0.641 0.727 0.432 0.432
> plotTree(tree)
> edgelabels(tree$edge.length)
Thank you, this was a great help.
ReplyDeleteThanks Liam, just used this snippet.
ReplyDeleteHey Liam!
ReplyDeleteI got here through googling, and was trying out your code on a tree with 100K tips. It's a little slow because of the sapply, so I looked for a faster solution. I found a faster solution here: https://rdrr.io/cran/paleotree/src/R/modifyTerminalBranches.R
```
branchSel <- tree$edge[,2]<(Ntip(tree)+1)
ee = tree$edge.length[branchSel]
```
although in this case `ee` does not have names that correspond to the tips, but it still does what the OP asked for and is a lot quicker on *very* large trees.